近日,我室周益装博士在国际期刊BMC Genomics在线发表了题为《A high-resolution genomic composition-based method with the ability to distinguish similar bacterial organisms》的研究论文。该研究报道了一种新的基于基因组组分的算法TZMD (Tetranucleotide-derived Z-value Manhattan Distance)。
基因组组分是指基因组上寡核苷酸(2-9个核苷酸)的频率,具有物种特异性,能够用来区分细菌物种。四联核苷酸能够平衡物种区分的准确度和计算机消耗,因而被广泛运用。该论文首先比较了目前已经报道的四种四联核苷酸的统计方法,发现TETRA (tetranucleotide-derived z-value Pearson correlation coefficient)能够代表其它所有方法。但是,早期的研究以及本研究都发现,TETRA不能区分相似物种以及种内菌株。该论文首先分析了造成这种低区分度的原因,发现源于TETRA的低分辨率。因此,研究人员提出了一个基于曼哈顿距离的新算法TZMD,并且从理论和实际数据两个方面都证明TZMD具有相比TETRA更高的分辨率。
TZMD不仅改善了基于基因组组分的物种区分效果,而且能够区分来自同一个基因型种的相似物种、亚种和种内菌株(如下图)。此外,该论文还发现,TZMD=0是一个区分克隆与非克隆菌株的可靠性高且易用的指标,首次揭示基因组组分具有菌株特异性。这个算法将为细菌的菌株分型、相似菌株区分等提供技术支持。
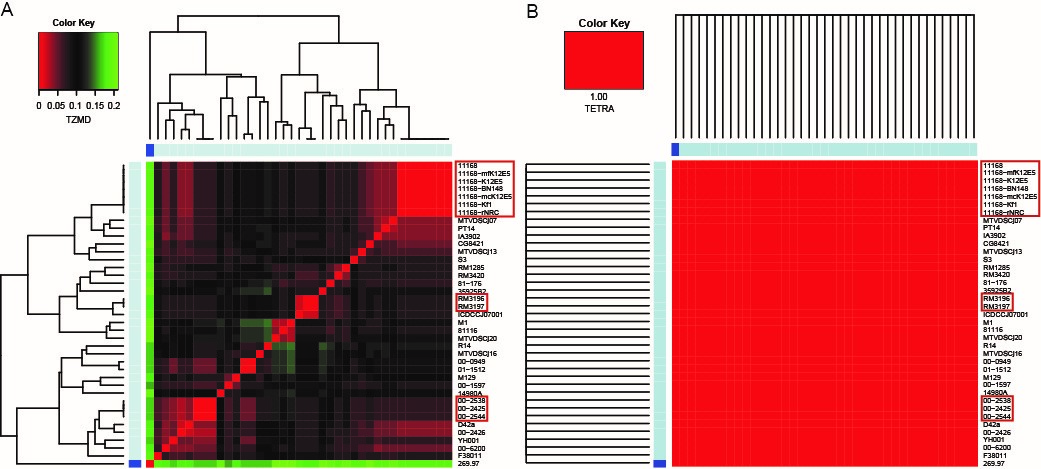
图A TZMD能够区分亚种和种内菌株;图B TETRA不能区分亚种和种内菌株
注:以空肠弯曲菌为例子,269.97是doylei亚种,其它为弯曲亚种。红色框标记,来自同一个克隆的菌株。
周益装博士为该论文的第一作者,周益装和其博士后合作导师金俊飞研究员为该论文的共同通讯作者。该研究得到了国家自然科学基金、漓江学者团队基金、广西科技基地与人才专项基金等多方面的资助。
论文链接:https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-019-6119-x
(文/钱颖超,审核/胡伟)